Predittori genetici di suscettibilità alle infezioni fungine cutanee: Uno studio pilota di associazione genome-wide, per affinare la ricerca di un gene candidato
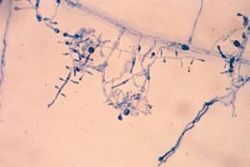 Trichophyton tonsurans è il patogeno fungino più importante nei bambini appartenenti alle minoranze negli U.S. Nonostante i tassi di infezione travolgenti, sembra che questo fungo infetta i bambini in modo specifico.
Trichophyton tonsurans è il patogeno fungino più importante nei bambini appartenenti alle minoranze negli U.S. Nonostante i tassi di infezione travolgenti, sembra che questo fungo infetta i bambini in modo specifico.
OBBIETTIVO
Questo studio è stato progettato per identificare i geni che possono predisporre o proteggere un bambino da un'infezione di T. tonsurans.
METODI
Sono stati candidati per l'inclusione, quei bambini partecipanti a uno studio longitudinale precedente, nel quale i tassi di infezione erano stati determinati in modo attendibile. Il DNA da un sottogruppo (n = 40) di questi bambini, agli estremi della popolazione, è stato sottoposto a genotipizzazione dell'intero genoma (WGG). Sono state esaminate le frequenze alleliche tra i casi e i controlli e sono stati utilizzati gli SNPs significativi per sviluppare un elenco del gene candidato per i quale è stato genotipizzato il resto della coorte (n = 115). È stato esaminato il tasso di infezione cumulativo a partire dal genotipo ed è stata esplorata la capacità dei genotipi selezionati di prevedere la probabilità di infezione, mediante l'analisi multivariata.
RISULTATI
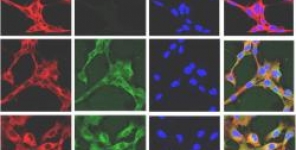
23 geni con un ruolo putativo meccanico nell'infezione cutanea sono stati selezionati per la valutazione. Di questi, 21 hanno dimostrato differenze significative nel tasso di infezione tra i genotipi. Un indice di rischio assegnato ai genotipi dei 21 geni ha giustificato oltre il 60% della variabilità osservata nel tasso di infezione (corretto r2 = 0.665, p < 0.001). Tra questi, 8 sembravano spiegare la maggior parte della variabilità che è stata osservata (r2 = 0.603, p < 0.001). I geni inclusi sono stati coinvolti: nell'attivazione e migrazione dei leucociti, nell'integrità e nel rimodellamento della matrice extracellulare, nella manutenzione dell'epidermide e nella riparazione delle ferite, e nella permeabilità cutanea.
CONCLUSIONI
Applicare WGG agli individui agli estremi del fenotipo può aiutare a guidare la selezione dei geni candidati nelle popolazioni di piccole coorti dove l'eziologia della malattia è probabilmente poligenica in natura.
Storia della pubblicazione:
Titolo: Genetic predictors of susceptibility to cutaneous fungal infections: A pilot genome wide association study to refine a candidate gene search
Rivista: Journal of Dermatological Science. doi:10.1016/j.jdermsci.2012.05.003
Autori: Susan M. Abdel-Rahman, Barry L. Preuett
Affiliazioni: Division of Pediatric Clinical Pharmacology and Medical Toxicology, The Children's Mercy Hospital and Clinics, Kansas City, MO, USA
Department of Pediatrics, University of Missouri-Kansas City, School of Medicine, Kansas City, MO, USA
Abstract:
Background
Trichophyton tonsurans is the foremost fungal pathogen of minority children in the U.S. Despite overwhelming infection rates, it does not appear that this fungus infects children in a non-specific manner.
Objective
This study was designed to identify genes that may predispose or protect a child from T. tonsurans infection.
Methods
Children participating in an earlier longitudinal study wherein infection rates could be reliably determined were eligible for inclusion. DNA from a subset (n = 40) of these children at the population extremes underwent whole genome genotyping (WGG). Allele frequencies between cases and controls were examined and significant SNPs were used to develop a candidate gene list for which the remainder of the cohort (n = 115) were genotyped. Cumulative infection rate was examined by genotype and the ability of selected genotypes to predict the likelihood of infection explored by multivariable analysis.
Results
23 genes with a putative mechanistic role in cutaneous infection were selected for evaluation. Of these, 21 demonstrated significant differences in infection rate between genotypes. A risk index assigned to genotypes in the 21 genes accounted for over 60% of the variability observed in infection rate (adjusted r2 = 0.665, p < 0.001). Among these, 8 appeared to account for the majority of variability that was observed (r2 = 0.603, p < 0.001). These included genes involved in: leukocyte activation and migration, extracellular matrix integrity and remodeling, epidermal maintenance and wound repair, and cutaneous permeability.
Conclusions
Applying WGG to individuals at the extremes of phenotype can help to guide the selection of candidate genes in populations of small cohorts where disease etiology is likely polygenic in nature.